RIEM : Dossier d'enquête
Épistémologie ● Philosophie des Sciences ● Histoire des Sciences
Édition, 20 p., A7
Marie Jammet
Designeuse graphique
@jamaismarie
http://jamaismarie.com/
Adrien Foucart
Chercheur post-doctorant
LISA – École polytechnique de Bruxelles – ULB
https://adfoucart.be/
Arthur Elskens
Doctorant
LISA – École polytechnique de Bruxelles – ULB
https://github.com/aelskens

























Fusion d’images médicales
« précliniques » multimodales
Fig. 1. Exemple d’imagerie CT in-vivo d’une souris : coupes selon les plans anatomiques et visualisation 3D du squelette. La croix marque la position d’une tumeur du foie.
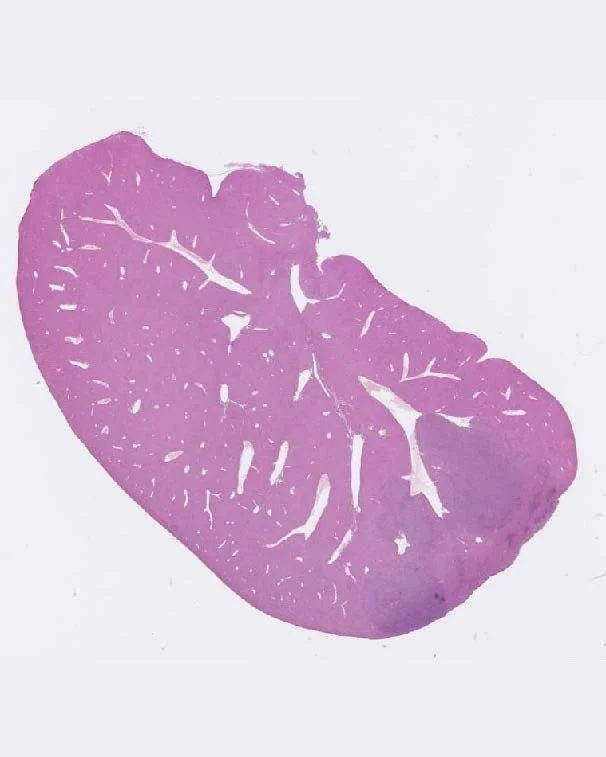
Fig. 2. Exemple d’imagerie au microscope d’une coupe du lobe gauche du foie contenant la tumeur (zone un peu plus foncée en bas à droite
Lorsqu’on réalise des expériences « précliniques » de traitement (c’est-à-dire sur des animaux, généralement des souris), on cherche à observer l’évolution de la maladie ainsi que l’effet du traitement sur l’organisme sur toute la période du traitement. Ces observations impliquent différentes modalités d’imagerie :
Des CT scans, où l’on voit bien les os et l’anatomie globale, mais mal les contrastes au sein des tissus (Fig. 1).
Des IRM qui permettent de mieux voir les tissus, et de mettre en avant certains phénomènes physiologiques.
De l’imagerie nucléaire (PET / SPECT), qui permet de cibler très spécifiquement ces phénomènes physiologiques, mais avec une mauvaise résolution spatiale.
De l’imagerie au microscope des tissus ex-vivo (c’est-à-dire après les avoir extraits de l’animal), qui permettent de mettre en avant des effets à l’échelle cellulaire (Fig. 2).
Idéalement, on voudrait pouvoir fusionner toutes ces informations pour se faire la meilleure idée possible de ce qu’il se passe durant le traitement. Mais d’une image à l’autre…
La nature des informations visibles change.
La résolution spatiale change.
Les objets étudiés changent (position / forme des organes évolue au cours du temps)
La fusion de ces images permet par exemple de pouvoir valider que certains phénomènes visibles au microscope sont détectables in-vivo via l’imagerie nucléaire, ou via des séquences IRM particulières (IRM quantitative), ce qui permettrait ensuite de réduire le nombre d’animaux nécessaires pour mener l’expérience (si on ne peut mesurer certains paramètres que ex-vivo, on doit sacrifier des animaux à plusieurs moments durant le traitement pour mesurer les évolutions).
Dans ce projet, nous appliquons des techniques d’analyse d’image et d’intelligence artificielle pour essayer d’automatiser autant que possible le processus de « recalage », qui permet de trouver la transformation (rotation, translation, déformation locale, mise à l’échelle…) à appliquer sur une image pour l’aligner sur une autre. Ces recalages peuvent être basés sur la détection de points ou de structures de référence particulières visibles sur les différentes modalités (par exemple : les vaisseaux sanguins), et/ou sur des techniques recherchant les corrélations entre les informations présentes dans les différents types d’imagerie.